独立行政法人理化学研究所(野依良治理事長)は、独自に開発してきた転写開始点同定技術「Cap Analysis of Gene Expression(CAGE)法※1」を1分子シーケンサー「Helicos® Genetic Analysis System※2」に適用し、わずか100 ナノグラムの RNA サンプルから、定量的かつ高精度に遺伝子発現量を測定することができる「HeliScopeCAGE法」を開発しました。これは、理研オミックス基盤研究領域(理研OSC、林崎良英領域長)と米国ヘリコスバイオサイエンス社(Helicos BioScience社)※3との共同研究の成果です。
生命現象を分子レベルで解明するには、多様な細胞の特徴をそれぞれ理解する必要があります。例えば、がん細胞とその近くに存在する正常細胞の遺伝子発現を比較することができると、がん細胞を定義し、医療に役立てることが可能となります。そのためには、少数の純粋な細胞から、微量のサンプルを抽出して解析する技術が必要です。しかし、これまでのシーケンサーでは、ポリメラーゼ連鎖反応(PCR法※4)によるDNA増幅工程が欠かせず、この工程中に遺伝子発現解析データの偏り(バイアス)が生じてしまうため、少量のサンプルから高精度な解析を行うことは不可能でした。
研究グループは、理研が独自開発した遺伝子解析技術である CAGE 法を、DNA増幅を使わずに1分子レベルで核酸の塩基配列を解読(シーケンス)することができるHeliScopeTM向けに最適化した、「HeliScopeCAGE法」を開発しました。この方法により、微量サンプルの直接シーケンスが可能となり、バイアスの削減とデータ再現性の向上を実現しました。バイアスが削減したために測定感度も改善し、わずか100ナノグラムのRNAサンプルから、遺伝子発現の定量解析を行うことが可能となりました。遺伝子発現解析によく使われるマイクロアレイ法との相関を確認するため、HeliScopeCAGE法とマイクロアレイ法を用いて、ヒト急性単球性白血病由来のTHP-1細胞株とヒト子宮頸がん由来のHeLa細胞株の、発現に差があった遺伝子数を検出したところ、従来のシーケンス法では見られなかった高い相関性が確認されました。
今後は、この高精度な技術を利用して、微量のサンプルから、従来の方法では検出できなかった微細な細胞特異的遺伝子発現の検出が可能となります。理研OSCは、多くの研究者が高精度な遺伝子発現解析を実施できるよう、日本のシーケンス拠点※5としての役割を担い、開発した技術を広く提供していきます。
この成果は、米国の科学雑誌『Genome Research』に掲載されるに先立ち、5月19日 にオンライン掲載されます。
- 背景
2003年4月に「ヒトゲノム計画」が完了し、ヒトゲノムの全塩基配列の解読が宣言されました。それ以来、ヒトをはじめとするさまざまな生物を対象にしたゲノムシーケンスの目的と手法は、多様化の一途をたどっています。これまでは、シーケンス処理能力(スループット)が注目されていましたが、現在では、例えばがんなどの疾病の診断に向けたシーケンス技術開発が進んでいます。これまでの歴史を振り返ってみると、20世紀末から21世紀初頭にかけては、第一世代シーケンサーである「蛍光キャピラリーシーケンサー※6」により、ヒトゲノムをはじめとする多数の生物のゲノムが決定されました。2005年になると、DNAポリメラーゼなどによるDNA合成法を用いた蛍光・発光などを検出して、超並列的な塩基配列決定を可能にする次世代シーケンサーが登場しました。現在の主流は、 逐次 DNA 合成・光検出法による超並列シーケンス手法を組み込んだ第二世代シーケンサーですが、2008年には、第三世代シーケンサーである「1分子シーケンサー」が市場に登場し、注目を集めています。
最先端のシーケンサーで解析する際には、すでに使用方法が確立したシーケンサーを使用する場合と異なり試行錯誤を伴います。機器の物理的な動作確認や調整に始まり、用途ごとにサンプルを調製する技術、従来経験したことのない膨大なデータの処理、それらの中から有用な情報を取り出すためのバイオインフォマティックス技術の開発などが欠かせません。
理研 OSCでは、世界をリードするゲノム・トランスクリプトーム(RNA)解析の研究活動を通して蓄積してきた最先端技術とノウハウを活かし、2009 年4月以降、日本のシーケンス拠点として活動しています。約20台の多種多様なシーケンサーを導入し、研究目的に合ったサンプル調製技術やバイオインフォマティックス技術を統合してスケールメリットを持たせるとともに、さらなる技術の開発と普及に務めています。特に1分子シーケンサーHeliScopeTMは、1分子のDNAを直接解析することができるため、従来のシーケンサーでは不可能だった細胞1個を解析する技術開発に向けて重要な役割を担うと考えています。そこで2008年、米国ヘリコスバイオサイエンス社との共同研究を開始し、OSC が独自開発した遺伝子解析技術である CAGE 法をHeliScopeTM 仕様に適用する「HeliScopeCAGE法」の開発に取り組んできました。
- 研究手法と成果
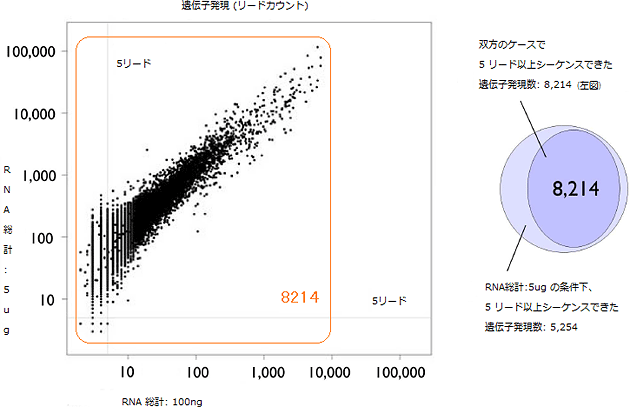
研究グループは、理研が独自開発した遺伝子解析技術CAGE法を、DNA増幅工程を不要とするHeliScopeTMに適合させることにより、従来のCAGE法で必要であった、二本鎖cDNA合成工程やDNA増幅工程を省き、よりシンプルな「HeliScopeCAGE法」の手順を確立することに成功しました(図1)。その結果、DNA 増幅処理で生じていたデータの偏り(バイアス)を回避し、高いデータの再現性を達成しました。実際に、ヒト急性単球性白血病由来のTHP-1細胞株を用いて、RNAの遺伝子発現の定量解析を行い、同じ検体から作製した2セットのサンプルの測定データを比較したところ、ピアソンの積率相関係数が0.989(1に近いほど正の相関がある)と非常に高い再現性を示しました(図2)。また、測定感度が高まり、ダイナミックレンジ(識別可能な範囲)の幅が従来技術(3 桁程度)に比べて5 桁以上と大きく広がるだけでなく(図2)、わずか100 ナノグラムの RNA サンプルから遺伝子発現量を定量化することに成功しました(図3)。
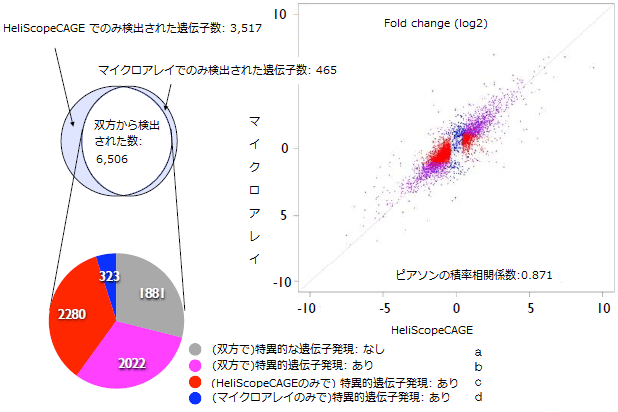
また、THP-1およびHeLa細胞株における遺伝子発現の違いを、HeliScopeCAGE法とマイクロアレイ法で解析した結果、マイクロアレイ法との高い相関を得ただけでなく、発現に差があった遺伝子の数を比較すると、HeliScopeCAGE法では、マイクロアレイ法より1,957個も多い4,302個の遺伝子発現の差を検出し、その高い精度と広い解析範囲を証明しました(図4)。
- 今後の期待
現在、バイオ医学研究分野を中心として、インフォマティクス手法の開発や次世代シーケンサーの高度化を目指した競争が世界中で激化しています。今回開発したHeliScopeCAGE法により、100ナノグラムという少量のサンプルから、PCR法によるバイアスのない、より精確な遺伝子発現解析が可能になりました。この技術を使うことにより、現在の技術では不可能な、細胞1個の遺伝子発現の解析が可能になると期待されます。個々の細胞における遺伝子発現ネットワークを解析し、細胞の機能を定義することができるようになると、例えば、がん細胞のように疾患に関連する細胞を迅速に発見することが可能になります。
OSCでは、HeliScopeCAGE法を駆使して、遺伝子発現ネットワーク解析技術をさまざまな細胞へ応用し、ヒト細胞の多様性の解明に取り組んでいます。これらの技術と研究成果を、がん治療や再生医療などの医療応用に役立てていきます。
| ※1 | Cap Analysis of Gene Expression(CAGE)法 |
| 理研が開発した方法で、耐熱性逆転写酵素やcap-trapper法を組み合わせて、5'末端から20塩基のタグ配列を切り出し、塩基配列を決定する実験技法。この塩基配列を読み取ってゲノム配列と照らし合わせ、どの部分がコピーされているかを調べることができる。 | |
| ※2 | Helicos® Genetic Analysis System |
| tSMSTMTM(true Single Molecule Sequencing)技術の開発により、わずか1分子のDNAを鋳型として、増幅することなくシーケンスできる特徴を持つ。一度にシーケンスできるDNAの長さは30塩基前後。高度に並列化されているため、一度の操作で約4億塩基を読むことができるのも際立った特徴の1つ。 | |
| ※3 | ヘリコスバイオサイエンス社 (Helicos BioSciences社) |
| tSMSTM (true Single Molecule Sequencing) 技術を中心とする、革新的なDNAシーケンス技術を開発する米国企業。米国が中心となって進めている1,000ドルゲノム計画(1,000ドルで1人のゲノムを読めるようにしようという計画)の資金提供を受けて2004年に設立。 | |
| ※4 | PCR 法 |
| ポリメラーゼ連鎖反応によるDNA増幅法。わずかな量の既知配列を持つDNAから、数十万倍に増幅できる。これを利用して、知りたい遺伝子の発現の有無及び量を増幅して検出することができる。 | |
| ※5 | シーケンス拠点 |
| 性能や用途が多様化する、いわゆる次世代シーケンサーの効果的活用法を開発し、その技術およびノウハウなどを広くライフサイエンス研究に供することを目的とする拠点。 OSCは、文部科学省革新的細胞解析研究プログラム(セルイノベーション)における「シーケンス拠点」に採択され、多様かつ大量のデータを取り扱うデータ解析拠点(国立遺伝学研究所)と緊密に連携して、先導研究を強力に支援している。(2009年4月23日プレス発表) | |
| ※6 | 蛍光キャピラリーシーケンサー |
| 毛細管(キャピラリー)内でDNAを分離し、蛍光標識で塩基配列を解析する装置。特殊な高分子の水溶液を充填した毛細管内で発揮する分子ふるい効果を利用して、試料中のDNAをその塩基数の順に分離することができる。 |
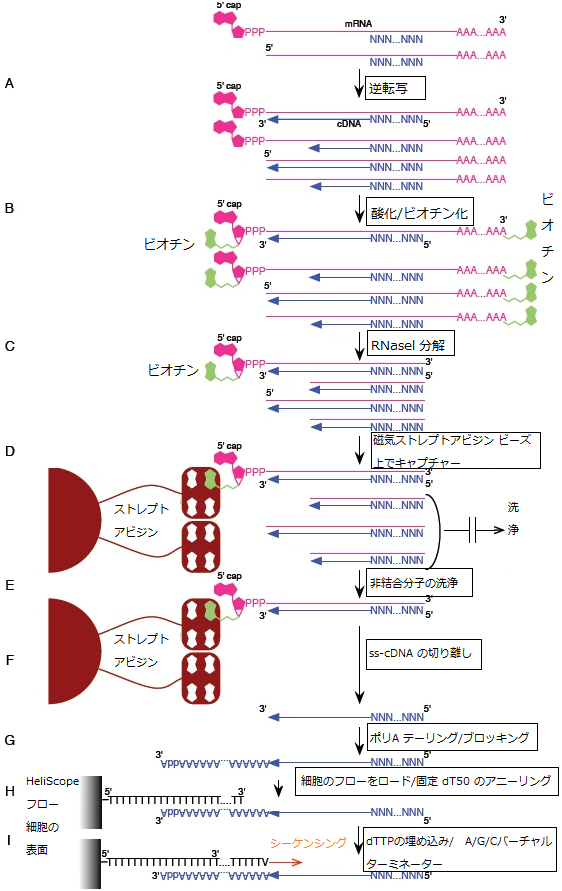 図1 HeliScopeCAGE法の手順
図1 HeliScopeCAGE法の手順
(A)逆転写し、cDNA を合成を合成
(B)CAP 構造を酸化してビオチン化
(C)一本鎖RNA部分をRNaseIで分解
(D)ビオチン化したRNA/cDNA混成分子を磁気ストレプトアビジンビーズ上で捕捉
(E)洗浄し、結合していないRNA/cDNA混成分子を取り除く
(F)一本鎖cDNAを切り離す
(G)切り離されたcDNAをポリAテール付加及び末端ブロッキング
(H)HeliScopeのフローに装着し、固相化されたdT50と二本鎖形成(アニーリング)
(I)はみ出したポリA部分をTTPで埋め、その末端をA/C/Gバーチャルターミネータで固定。
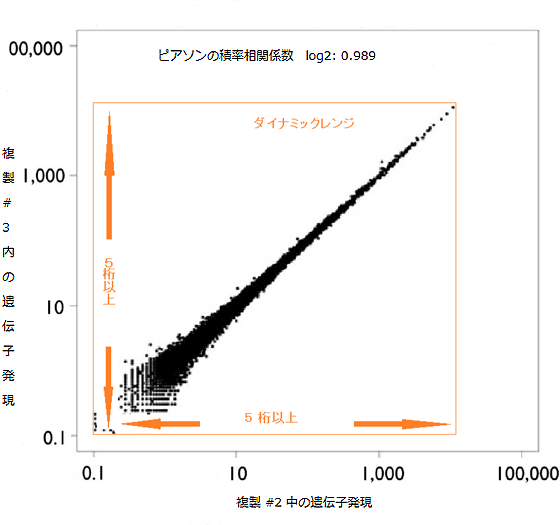
図2 異なるデータ間での遺伝子発現における再現性の相関
横軸は複製#2の、縦軸は複製#3の遺伝子発現を表す。両データの解析値がほぼ同一であることから、2回の測定を通じて高いデータ再現性を示していることが分かる。