ゲノム上の遺伝情報はmRNAに転写された後,タンパク質の設計情報として働きます。
mRNAの塩基配列にはタンパク質を構成するアミノ酸配列が直接コードされているため,タンパク質の機能を解析する上で非常に重要な情報です。マウスエンサイクロペディア計画ではマウスを材料として,様々な組織や細胞からこのmRNAの完全なコピーであるcDNAのクローンおよびその配列(完全長cDNAシーケンス)を網羅的に収集しました。ここでは、当研究グループが開発した一連の完全長cDNAライブラリ作成技術について概説します。これらの技術は、さまざまな生物種のcDNA解析に用いられています。
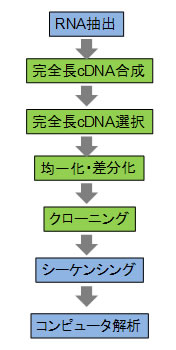
完全長cDNAプロジェクトの概略
技術の概要
- 伸長法(トレハロース法)[1]
従来、逆転写反応は、第一鎖cDNA合成の効率が悪いことが最大の問題でした。当研究グループでは、トレハロースを添加することで、この問題が克服できることを発見しました。トレハロースは二糖類で、ヒートショックをかけた酵母菌で誘導されることが知られていました。私たちはこれをヒントに、トレハロースが逆転写酵素を含む酵素に耐熱性を附与することを見出したのです。これにより、逆転写酵素を用いた反応を通常の42℃ではなく、60℃で行うことができるようになりました。60℃では、鋳型RNAの二次構造が弱まり、mRNAの5‘末端の非翻訳領域にしばしば存在する安定な二次構造をもった領域も効率的に逆転写していくことが可能になりました。 - 選択法(キャップ・トラップ法)[2] [3]
伸長反応が不完全に終わったcDNAをライブラリから除くために、ビオチン化キャップ・トラップ法を開発しました。
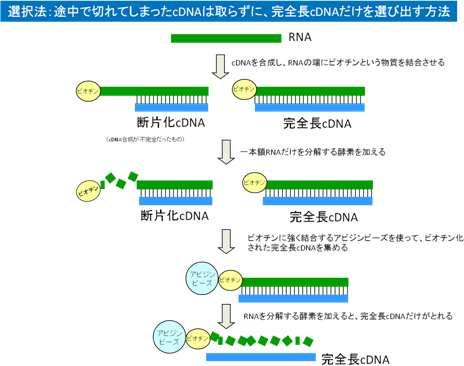
この方法では、はじめに真核生物のmRNAに特有のキャップ構造をビオチン化します。
ついで、RNaseI処理により、不完全長cDNAのビオチン化したキャップ構造だけを選択的に取り除きます。残ったビオチン化したキャップを有する完全長cDNAは、ストレプトアビジンをコートした磁気ビーズを用いることにより「釣り上げる」ことができます。このようにして選択された完全長cDNAをアルカリ処理することにより、ビーズから溶出します。次に、ビーズから分離され1本鎖となった第一鎖cDNAの第二鎖を合成することにより、完全長cDNAだけを選択的に得ることができます。 - 新規クローニングベクター[4]
従来、完全長cDNAライブラリの作成においては、「クローニングバイアス」という問題がありました。つまり、mRNAの長さが短いクローンの方がクローニングされやすく、短いクローンの割合が増えてしまうのです。これは、トレハロースの存在下でも短いcDNAの方が長いcDNAよりも生成しやすいことに加えて、一般的なベクターは短いcDNAをクローニングしやすい傾向があることが原因でした。
そこで、当研究グループでは、通常クローン化が可能とされている6kb~20 kb のcDNA をカバーできる新規ベクターの構築に取り組みました。この新規ベクターはλFlcIII-L と名付けられ,平均鎖長はこれまでのcDNA ライブラリの中でも最長の6.9 kb となりました。このベクターを用いることにより,従来の方法では難しかった長鎖cDNA の単離,シーケンス決定を行うことができるようになりました。次に,このλFlcIII-L ベクターを更にバックグランドの低減とシーケンス決定後の機能解析への利用を考えて改良し,λFlcIVというベクターを作成し、これを利用することにより、バックグランドがゼロに近いcDNA ライブラリを作成することが可能となりました。
- 均一化・差分化法[5]
細胞や組織から得られるmRNAは発現量の多い1,500種類程度のmRNAが全RNAの50~60%を占めています。このため、非常に発現量の少ないcDNAを1回のシーケンスで同定しようとすると、同定効率が著しく低くなってしまいます。これは、発現しているすべてのRNAを網羅的に同定しようとするプロジェクトでは大きな問題となりました。 そこで、ランダムシーケンス解析の効率を上げるため、ライブラリ中で発現頻度の高いcDNAが占める割合を減らすことが必要になります。極端にいえば、すでに同定したcDNAはすべてライブラリから除いてもよいのです。このための方法として、ライブラリ中のcDNAの頻度を均等にする「cDNA均一化」、および目的としないcDNAをライブラリから取り除く「差分化」法を確立しました。
一本鎖核酸が相補鎖と再会合する際の動態は、単純には濃度の高い核酸は濃度の低い核酸よりも早く再会合します。この現象を利用して、均一化および差分化を行います。
まず、cDNAを調製するときに用いたmRNAの残りをビオチン化します。これをドライバーといいます。ついで、第一鎖cDNA(一本鎖DNAであり、mRNAとは相補的配列でテスターとよびます)と同量のドライバーを混ぜます。そして、適切なコントロール下でテスターとドライバーを再会合させます。この条件で、mRNAと再会合したcDNA(主に高頻度、中頻度のものが再会合するようにコントロールする)は、ビオチン化したmRNAが結合しているため、磁気ビーズで選択的に取り除くことができます。
応用
一連のcDNA技術は、ごく少量の組織にも適用できるようプロトコルに改良を加えるなどして、さまざまな生物種に適用されています。
キャップ・トラップ法の応用技術として、遺伝子転写産物の5’末端をタグとして切り出すCAGE法を開発ました。これらの方法を活用して、「RNA新大陸の発見」などの画期的な成果をあげてきました。
参考文献
- ^ P. Carninci, Y. Nishiyama, A. Westover, M. Itoh, S. Nagaoka, N. Sasaki, Y. Okazaki, M. Muramatsu, and Y. Hayashizaki, Thermostabilization and thermoactivation of thermolabile enzymes by trehalose and its application for the synthesis of full length cDNA, Proc Natl Acad Sci U S A 95, 520-524 (1998), doi: 10.1073/pnas.95.2.520
- ^ P. Carninci, C. Kvam, A. Kitamura, T. Ohsumi, Y. Okazaki, M. Itoh, M. Kamiya, K. Shibata, N. Sasaki, M. Izawa, M. Muramatsu, Y. Hayashizaki, and C. Schneider, High-efficiency full-length cDNA cloning by biotinylated CAP trapper, Genomics 37, 327-336 (1996), doi; 10.1006/geno.1996.0567
- ^ P. Carninci, A. Westover, Y. Nishiyama, T. Ohsumi, M. Itoh, S. Nagaoka, N. Sasaki, Y. Okazaki, M. Muramatsu, C. Schneider, and Y. Hayashizaki, High efficiency selection of full-length cDNA by improved biotinylated cap trapper, DNA Res 4, 61-66 (1997), doi: 10.1093/dnares/4.1.61
- ^ P. Carninci, Y. Shibata, N. Hayatsu, M. Itoh, T. Shiraki, T. Hirozane, A. Watahiki, K. Shibata, H. Konno, M. Muramatsu, and Y. Hayashizaki, Balanced-size and long-size cloning of full-length, cap-trapped cDNAs into vectors of the novel lambda-FLC family allows enhanced gene discovery rate and functional analysis, Genomics 77, 79-90 (2001), doi: 10.1006/geno.2001.6601
- ^ P. Carninci, Y. Shibata, N. Hayatsu, Y. Sugahara, K. Shibata, M. Itoh, H. Konno, Y. Okazaki, M. Muramatsu, and Y. Hayashizaki, Normalization and subtraction of cap-trapper-selected cDNAs to prepare full-length cDNA libraries for rapid discovery of new genes, Genome Res 10, 1617-1630 (2000), doi: 10.1006/geno.2001.6601
