nanoCAGE法は、従来のCAGE法がマイクログラムレベルのRNAサンプルを必要とするのに対し、その1000分の1のナノグラムレベルのRNAサンプルからの、ゲノムワイドなプロモーター活性定量解析を可能にしました。具体的には、rRNAやtRNAなどを含めたすべての種類のRNAのうち、mRNAからのcDNAだけを特異的に逆転写することが可能なテンプレートスイッチング法と、この方法で逆転写したcDNAだけを増幅するセミサプレッシブPCR反応を導入しました。その結果、ナノグラムレベルという非常に少ない量のmRNAから5´末端の25塩基のタグ配列を切り出し、次世代シーケンサーを用いて大規模に転写開始点を決定することができるnanoCAGE法の開発に成功しました( 図1)。この技術により、生体内に少数しかない細胞、例えば、神経細胞やがん細胞などの発現する微量なRNAの解析が可能になりました。
一方CAGEscan法は、微量なRNAを解析できる上に、次世代シーケンサーの能力を活用し、5´末端とその反対側である3´側の塩基配列の両方を同時に解析できるというメリットがあります。この技術により、RNAの5´末端が、そのRNA自体やほかのRNAの3´側の塩基配列に結合する様子を明らかにし、RNA間の相互作用を理解することができます。
これら2つの新しい技術により、微量サンプルからの詳細な遺伝子発現解析と効率的なRNA研究が可能となりました。また、薬物感受性解析などの医療分野や、全転写産物間相互作用解析などへの応用が可能です。
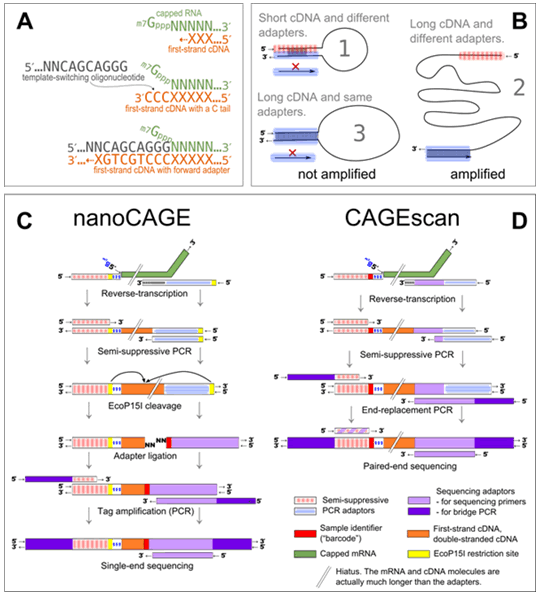
図1 : nanoCAGEおよびCAGEscanの実験プロトコール
(a) nanoCAGE法は、5'末端 をとらえるためにテンプレートスイッチングを利用する。5´末端にCAP構造(m7G)を持つRNA(緑)がcDNA(オレンジ)に逆転写されるとき(上段)、逆転写酵素はCAP構造の相補鎖となる部位にシトシン(C)を付加する(中段)。この付加されたシトシンは、CAP構造と疑似的な配列を持つリボグアノシンテンプレートスイッチングプライマー(黒)と相補鎖を作る。その結果、cDNAの両末端は違う配列が増幅される(下段)。この時点では長さの調整はできず、150塩基以下の短いものも、それ以上の長いものも逆転写される。長さの調整はBのセミサプレッシブPCR反応で行われる。
(b) セミサプレッシブPCR反応。150塩基以下の短いcDNAテンプレートは、末端の塩基同士が容易に相補鎖を形成するため(1)、プライマーがcDNAテンプレートと相補鎖を形成できずPCR反応が進まない。また、150塩基以上のcDNAテンプレートでも、逆転写反応中に両末端がテンプレートスイッチングオリゴヌクレオチドに置換されてしまったもの(3)はプライマーが相補鎖を形成できず、PCR反応が進まない。テンプレートスイッチング法で相補鎖を形成したcDNAテンプレート(逆転写反応で末端が別々のプライマーに置換された150塩基以上のcDNAテンプレート)だけがPCR反応で増幅される(2)。
(c) nanoCAGEタグの調製。テンプレートスイッチング反応とセミサプレッシブPCR反応後、EcoP15I酵素で切断した25塩基のタグに、バーコードを含むオリゴヌクレオチドアダプターを付加する。PCRで増幅した後、シーケンシングする。
(d) CAGEscan法。nanoCAGE 法と同様、テンプレートスイッチング反応とセミサプレッシブPCR反応を行う。その後、EcoP15I酵素で切断することなく、次世代シーケンサーIllumina Genome Analyzerで、5´末端と3´側の両方をペアエンド解析する。
参考文献
- Plessy C, et al. Linking promoters to functional transcripts in small samples with nanoCAGE and CAGEscan. Nature Methods 7 528–34 (2010), doi: 10.1038/nmeth.1470
- Md Salimullah, Mizuho Sakai, Charles Plessy and Piero Carninci. NanoCAGE: a high-resolution technique to discover and interrogate cell transcriptomes. Cold Spring Harbor Protocols 2011, doi: 10.1101/pdb.prot5559
- Tang DT, Plessy C, Salimullah M, Suzuki AM, Calligaris R, Gustincich S, Carninci P. Suppression of artifacts and barcode bias in high-throughput transcriptome analyses utilizing template switching. Nucleic Acids Res. 2013 Feb 1;41(3):e44. doi: 10.1093/nar/gks1128
- Harbers M, Kato S, de Hoon M, Hayashizaki Y, Carninci P, Plessy C. Comparison of RNA- or LNA-hybrid oligonucleotides in template-switching reactions for high-speed sequencing library preparation. BMC Genomics. 2013 Sep 30;14(1):665. doi:10.1186/1471-2164-14-665
